搜索


近日,EON体育4平台🎛、微生物代謝國家重點實驗室的吳更教授團隊與武漢大學王連榮教授團隊合作,闡明了古菌DNA磷硫酰化修飾途徑中的重要蛋白DndE的結構和功能。該研究成果以“Structural and functional analysis of DndE involved in DNA phosphorothioation in the haloalkaliphilic archaea Natronorubrum bangense JCM10635”為題發表在《mBio》雜誌上。EON体育4平台吳更教授👨🏿🦲、武漢大學王連榮教授為通訊作者。這項研究第一次詳細闡明了一個古菌來源的、在DNA磷硫酰化修飾途徑中起重要作用的蛋白DndE的結構和功能,加深了人們對於DNA磷硫酰化修飾的認識和理解📎。
在原核生物如細菌和古菌中❇️🦸🏼♂️,DNA的主鏈骨架上的一個非橋聯氧原子被替換為硫原子,這種特殊的表觀遺傳修飾稱為DNA磷硫酰化修飾。已發現的對DNA進行磷硫酰化修飾的酶系統包括兩種🕘:I型的DndA-DndB-DndC-DndD-DndE,對雙鏈DNA進行硫修飾;和II型的SspA-SspB-SspC-SspD🪦,對單鏈DNA進行硫修飾👩🏻🏭。DndE屬於I型DNA磷硫酰化修飾系統。
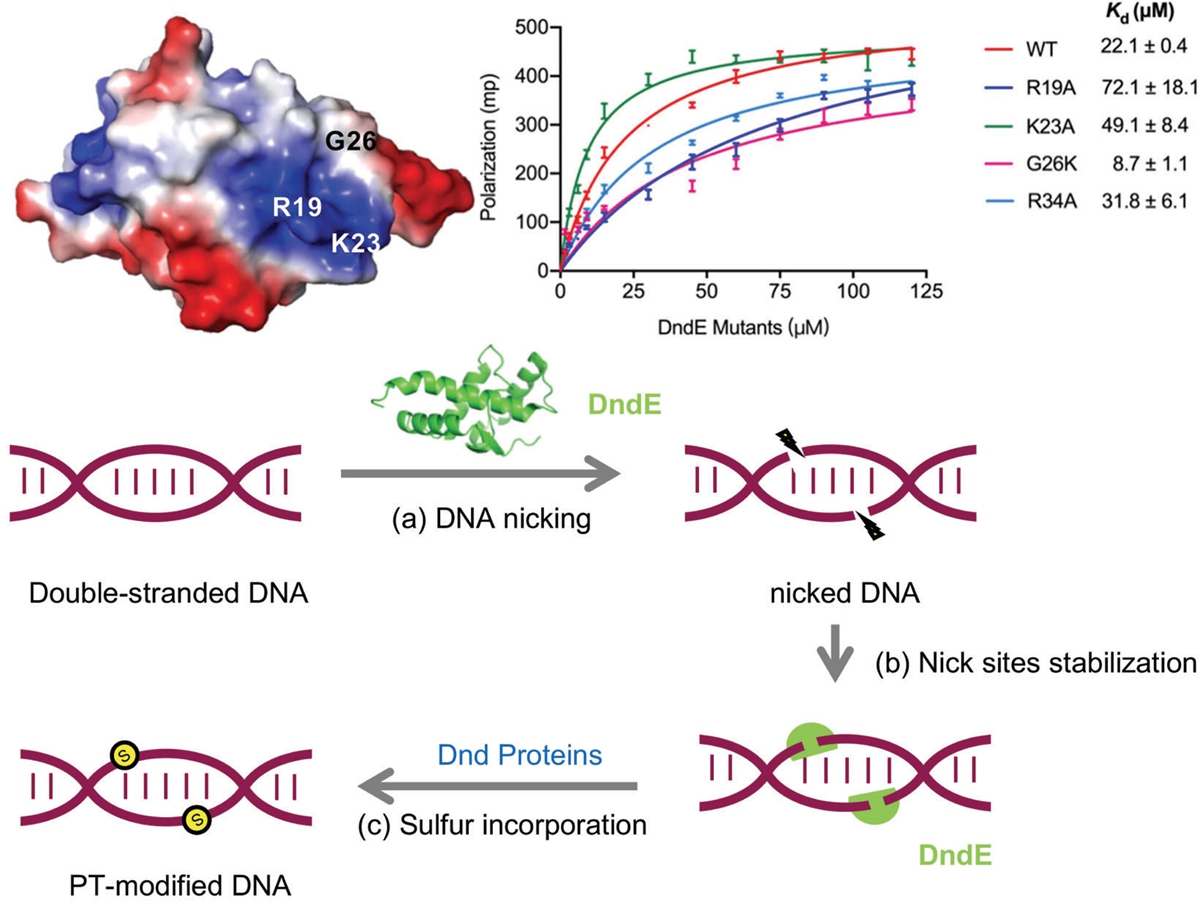
本研究首先通過解析嗜鹽堿古菌Natronorubrum bangense JCM10635菌株的DndE蛋白的晶體結構,發現與之前發表的大腸桿菌B7A菌株的DndE形成四聚體不同,古菌的DndE蛋白在晶體中和在溶液中都是以單體的形式存在。接著,本研究通過采用純化的DndE蛋白在體外對DNA的缺刻(nicking)實驗檢測發現,與II型DNA硫化修飾系統SspABCD中的SspB蛋白類似👩🏼🔬,DndE具有對DNA的缺刻活性,將共價閉環(covalently closed circular)雙鏈DNA的其中一條單鏈切斷🦉,即將DNA缺刻🐺,產生開環(open circular)DNA,並隨著時間的逐步推移🙅♀️,產生線狀(linear)DNA。而且,本研究還揭示🉐,DndE的這種對DNA的缺刻活性依賴於二價金屬陽離子🤢,特別是鎂離子,而不依賴於DNA上存在或不存在磷硫酰化修飾。Run-off測序結果顯示,DndE對DNA的缺刻活性也沒有DNA序列特異性🍈。然後✌🏽,通過采用在體外的熒光標記的DNA與純化的DndE蛋白的熒光偏振檢測實驗表明,DndE蛋白可以在體外與DNA直接結合,而且😯◀️,DndE與缺刻DNA的親和力最強🧑🌾,與雙鏈DNA的親和力次之👊🏿,與單鏈DNA的親和力最弱。最後,本研究發現將DndE蛋白表面帶正電荷的精氨酸19、賴氨酸K23和精氨酸R34突變為電中性的丙氨酸的話,會降低DndE與缺刻DNA之間的親和力☪️;而如果將DndE蛋白的甘氨酸26突變為帶正電荷的賴氨酸的話💸,會增強DndE與缺刻DNA之間的親和力。這說明DndE蛋白通過其表面帶正電荷的氨基酸殘基如精氨酸19、賴氨酸K23和精氨酸R34等來結合缺刻DNA🙎🏿♂️。
本文是吳更團隊自2018年Nature Communications上發表的細菌采用SBD結構域識別DNA上的硫化修飾的結構機理👩🏽🚒、2020年Nature Microbiology上發表的II型DNA硫化修飾系統的SspB和SspE晶體結構、2020年mBio上發表的II型DNA硫化修飾系統的半胱氨酸脫硫酶SspA晶體結構的延續和擴展👨🏼🦱。
論文鏈接:
https://pubmed.ncbi.nlm.nih.gov/35420474/


 首頁
首頁

 EON4概況
EON4概況

 師資隊伍
師資隊伍

 人才培養
人才培養

 招生就業
招生就業

 科學研究
科學研究

 平臺基地
平臺基地

 黨群工作
黨群工作

 校友之家
校友之家

 安全工作
安全工作

 網上辦事
網上辦事

 當前位置:
當前位置: